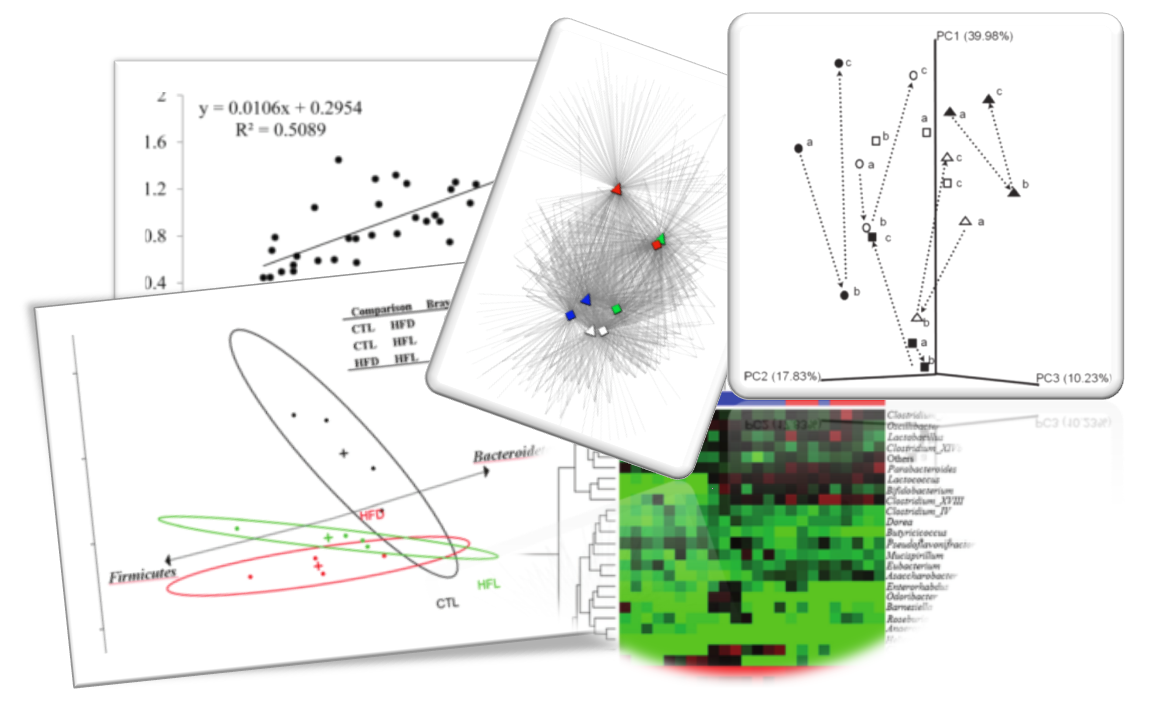
1차 PCR
준비물 : 96well PCR plate, PCR plate seal, V4 Primer (Forward, Reverse), KAPA HiFi Hot Start Ready Mix
- Primer와 KAPA HiFi를 용량에 맞게 5ml tube에 Mixture제작.
샘플 1개 기준
Forward Primer (10 pmol) : 5ul
Reverse Primer (10 pmol) : 5ul
KAPA HiFi : 12.5ul
- 만들어 놓은 Mixture를 96well PCR plate에 분주.
- 5ng/ul로 맞춰놓은 DNA 를 Multi channel Pipet을 이용해 5ul 씩 분주.
- Plate Seal을 Plate에 부착. (들뜨지 않게 잘 밀착시키기)
- Spin down 진행.
- PCR Running
95℃ 3m, 25cycle of 95℃ 30s, 55℃ 30s, 72℃ 30s, 72℃ 5m, Hold at 12℃
준비물 : 96well cell culture plate 4개 , HiAccu Beads, 80% EtOH, 10mM Tris buffer, Magnetic Extractor & cover, Magnetic Ring stand
- 96well cell culture plate 1개에HiAccu Beads 20ul를 분주.
- 1차 PCR Product(25ul)를 Pipetting을 이용해 Beads 와 혼합.
- 5분간 상온에서
- Incubation을 하는 동안 2개의 Plate에 80% EtOH 100ul를 분주, 1개의 Plate에는 10mM Tris buffer 52.5ul분주.
- Magnetic Extractor에 cover를 씌우고 Beads만을 Binding 시켜 첫번째 EtOH Plate로 옮기고 Ring Stand 를 이용해 Extractor cover로 부터Beads를 분리.
- 30초간
- 다시 Extractor를 이용해 Bead를 두번째 EtOH Plate로 옮긴후 Ring Stand 를 이용해Extractor cover로 부터Beads를 분리.
- 30초간
- Extractor를 이용해 Beads를 Binding 시킨 후 상온에서 10분간 Air Dry 진행. (Beads의 색으로 확인 진갈색 ->연갈색)
- Air Dry가 완료된 Beads를 Tris Buffer Plate로 옮겨Ring Stand 를 이용해Extractor cover로 부터Beads를 분리.
- 2분간
- 최종적으로 Extractor를 이용해 Beads를 제거.
준비물 : 96well PCR plate, PCR plate seal, Illumina Index Primer(B set or Cset), KAPA HiFi Hot Start Ready Mix, DNAase Free Water
- PCR Plate에 다음과 같이 순서대로 분주.
DNAase Free water : 10ul
Index Primer Forward : 5ul
Index Primer Reverse : 5ul
1차 Clean-up product : 5ul
KAPA HiFi : 25ul
- Plate Seal을 Plate에 부착. (들뜨지 않게 잘 밀착시키기)
- Spin down 진행.
- PCR Running
95℃ 3m, 8cycle of 95℃ 30s, 55℃ 30s, 72℃ 30s, 72℃ 5m, Hold at 12℃
준비물 : 96well cell culture plate 4개 , HiAccu Beads, 80% EtOH, 10mM Tris buffer, Magnetic Extractor & cover, Magnetic Ring stand
- 96well cell culture plate 1개에HiAccu Beads 56ul를 분주.
- 1차 PCR Product(50ul)를 Pipetting을 이용해 Beads 와 혼합.
- 5분간 상온에서
- Incubation을 하는 동안 2개의 Plate에 80% EtOH 100ul를 분주, 1개의 Plate에는 10mM Tris buffer 70ul분주.
- Magnetic Extractor에 cover를 씌우고 Beads만을 Binding 시켜 첫번째 EtOH Plate로 옮기고 Ring Stand 를 이용해 Extractor cover로 부터Beads를 분리.
- 30초간
- 다시 Extractor를 이용해 Bead를 두번째 EtOH Plate로 옮긴후 Ring Stand 를 이용해Extractor cover로 부터Beads를 분리.
- 30초간
- Extractor를 이용해 Beads를 Binding 시킨 후 상온에서 10분간 Air Dry 진행. (Beads의 색으로 확인 진갈색 ->연갈색)
- Air Dry가 완료된 Beads를 Tris Buffer Plate로 옮겨Ring Stand 를 이용해Extractor cover로 부터Beads를 분리.
- 2분간
- 최종적으로 Extractor를 이용해 Beads를 제거.